栾天罡/翟筠秋团队基于核酸探针设计新理论开发单细胞DNA损伤分析方法
近年来,随着精准医学和环境健康研究的深入发展,核酸修复酶作为维持基因组完整性的关键分子,其检测和功能研究已成为癌细胞识别、环境毒理学和药物研发的重要领域。近期,赌博app-网络赌博软件 栾天罡教授、广州中医药大学翟筠秋副研究员相关团队围绕核酸修复酶监测工具的开发和应用开展了一系列研究工作,在国际权威期刊Advanced Science、Analytical Chemistry和TrAC-Trend Analytical Chemistry发表多项重要成果。
成果一:核酸探针分子识别理论模型的构建与高灵敏探针研发
核酸修复酶是指示环境污染物诱导DNA损伤的关键生物标志物,其精准检测对揭示污染物毒性机制至关重要。核酸荧光探针可以被设计以靶向特定的核酸修复酶,实现在活细胞中原位实时监测核酸修复酶,是目前核酸修复酶不可或缺的分析方法(TrAC-Trend Anal. Chem., 2024, 171, 117489.)。针对现有研究缺乏系统性探针设计理论的问题,团队创新性地整合MD模拟与MM/PBSA计算方法,建立了解旋酶WRN与核酸相互作用的序列特异性预测理论,定量阐明了不同解旋酶对核酸探针GC端识别的热力学选择性机制,为特异性探针设计提供了理论基础(Adv. Sci., 2024, 2413368)。针对污染物诱导细胞癌变过程中关键生物标志物miR-21和FEN1,设计了基于DNA四面体的多靶标分析方法,通过构建DNA局域逻辑电路,实现了miR-21和FEN1的特异性同步检测,并成功用于细胞不同状态下生物标志物的变化监测(Anal. Chem., 2025, 97, 6258-6297.)。
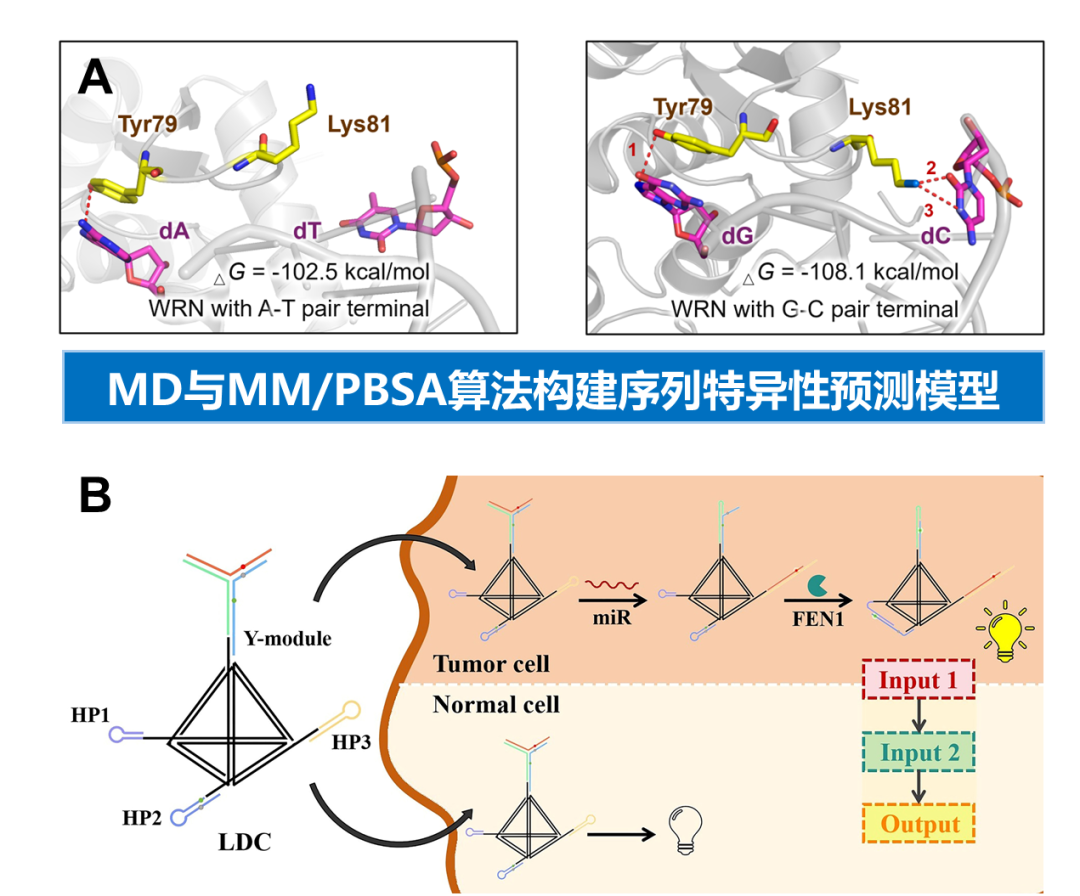
图1. (A) 核酸探针分子识别理论模型; (B) DNA局域逻辑电路实现双靶标同步检测
成果二:单细胞水平DNA损伤分析新方法与污染物毒性机制研究
环境污染物暴露会诱导细胞内多种类型DNA损伤,从而导致疾病的发生和发展。团队围绕核酸修复酶与DNA损伤的对应关系,发展了单细胞脱碱基损伤原位检测技术。制备了一种对脱嘌呤/脱嘧啶核酸内切酶1(APE1)高度敏感和特异性识别的核酸探针(ZnO2@DNA),用于指示活细胞中与APE1相关的DNA碱基损伤的位置和程度。研究首次发现苯并芘(BaP)可诱导脱碱基损伤,损伤程度分别与暴露时间(2-24 h)和暴露浓度(5-150 μM)呈正相关(Anal. Chem., 2023. 95(19). 7788-7795.)。进一步的,设计了双酶激活核酸探针,实现了碱基切除修复途径关键修复酶APE1和FEN1活性的同步、独立监测,成功解析了卤代多环芳烃6-Cl-BaP和甲基多环芳烃1-MP暴露后细胞群体中DNA损伤修复响应的异质性模式,发现6-Cl-BaP具有更强的细胞毒性,从单细胞水平阐明了污染物结构差异导致的毒性效应差异及其分子机制(Anal. Chem., 2025, 97, 4841-4849)。
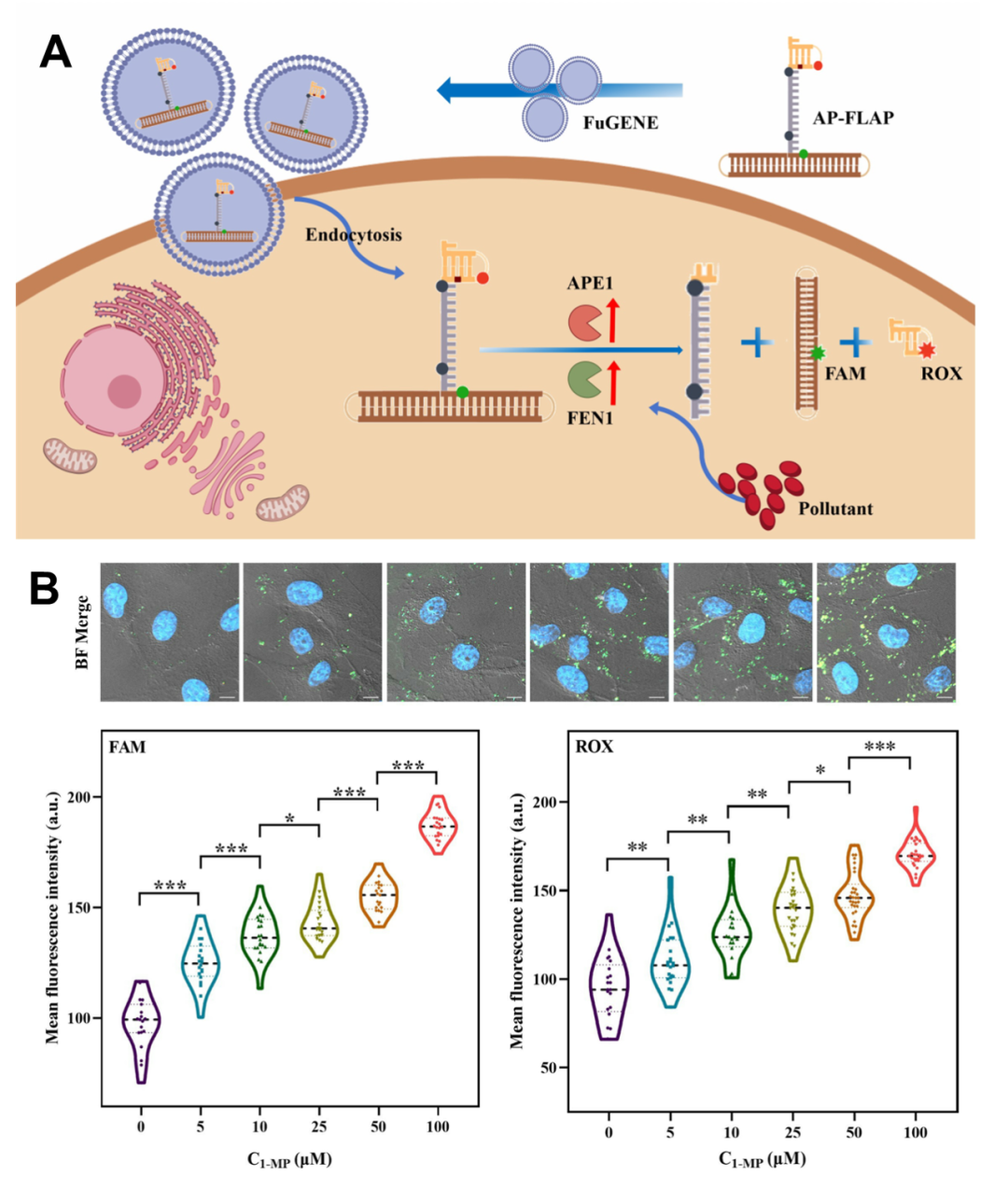
图2. (A) DNA损伤探针原理示意图;(B) 污染物暴露致DNA损伤及浓度相关性
成果三:高通量单细胞质谱代谢组学在环境毒理学中的应用
团队结合流式细胞术的高通量特性和质谱技术的组学优势,发展高通量单细胞质谱技术,用于分析单个原代肝细胞内的新型全氟化合物6:2 Cl-PFESA及其内源性代谢物。研究结果显示斑马鱼肝细胞对6:2 Cl-PFESA的积累存在显著的性别差异(p < 0.05),揭示了明显的性别依赖性代谢反应和6:2 Cl-PFESA引发的代谢失调,并强调了性别特异性的毒性反应。本研究证明了单细胞代谢组学在解析污染物暴露的细胞机制及代谢响应方面的可行性,为单细胞水平上的精准、全面毒性评估提供了新的见解(Anal. Chem., 2025, doi:
10.1021/acs.analchem.4c06345)。
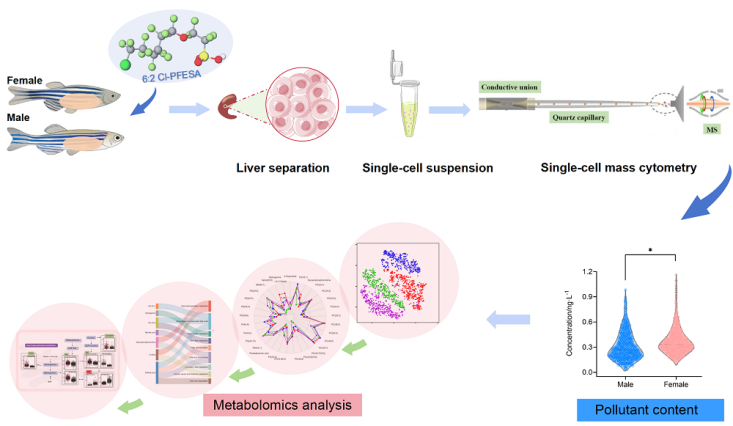
图3. 高通量单细胞质谱流式解析污染物暴露致肝细胞毒性
以上研究工作得到了国家自然科学基金重大科研仪器研制、广东省重点研发计划、国家自然科学基金青年基金项目等项目的支持。
论文链接
[1] TrAC-Trends in Analytical Chemistry, 2024, 171, 117489. DOI: 10.1016/j.trac.2023.117489.【成果一】
[2] Advanced Science, 2024, 2413368.DOI: 10.1002/advs.202413368.【成果一】
[3] Analytical Chemistry, 2025 97, 6258-6297. DOI: 10.1021/acs.analchem.5c00247.【成果一】
[4] Analytical Chemistry, 2023, 95(19). 7788-7795. DOI: 10.1021/acs.analchem.3c01251.【成果二】
[5] Analytical Chemistry, 2025, 97, 4841-4849. DOI: 10.1021/acs.analchem.4c03193. 【成果二】
[6] Analytical Chemistry, 2025, DOI: 10.1021/acs.analchem.4c06345.【成果三】



